Polymerase-Kettenreaktion
Die Polymerase-Kettenreaktion (Polymerase Chain Reaction, PCR) ist eine Methode, um Desoxyribonukleinsäure (DNA) zu vervielfältigen, ohne dafür lebende Organismen, wie z.B. Colibakterien oder Hefe zu verwenden. Eine umfassendere Darstellung findet sich auf Polymerase-Kettenreaktion (Langfassung).
Sie wurde Anfang der 1980er Jahre von Kary Mullis erfunden, wofür er den Nobelpreis für Chemie erhielt. 1989 ließen Hoffman La Roche und die Perkin-Elmer Corporation die PCR-Methode patentieren. PCR wird gewöhnlich in medizinischen und biologischen Forschungslaboren für eine Vielzahl von Aufgaben eingesetzt, unter anderem zur Erkennung von Erbkrankheiten, zur Identifikation genetischer Fingerabdrücke, zur Erzeugung von genetischen Stammbäumen, zur Klonierung von Genen (siehe unten) und für Vaterschaftstests.
PCR benötigt sehr wenig Ausgangsmaterial, in manchen Fällen genügt ein einziger DNA-Strang. Während der Kettenreaktion wird die DNA durch das Enzym DNA-Polymerase kopiert. Normalerweise wird nur ein kleiner Teil eines langen DNA-Strangs durch PCR verdoppelt. Dieser Teil wird durch die Primer, kurze, künstliche DNA-Stücke von etwa 20-40 Basenpaaren Länge (DNA besteht aus einem Doppelstrang, daher wird ihre Länge in komplementären Einheiten, den Basenpaaren, gemessen) festgelegt, welche genau mit dem Anfang bzw. dem Ende des zu kopierenden Strangs übereinstimmen.
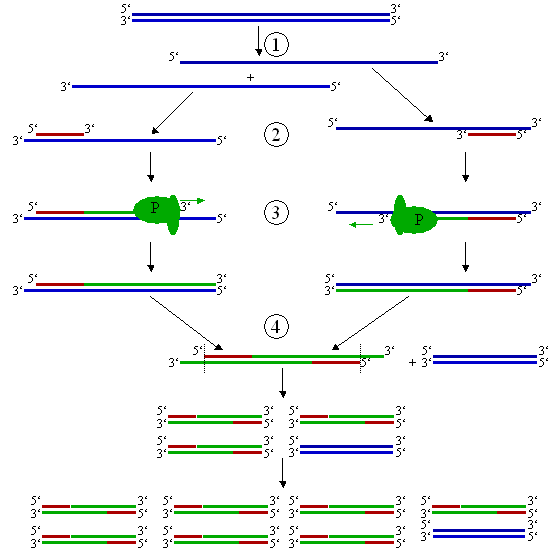
Die PCR-Maschine besteht aus einem computerkontrollierten Ofen mit Kühleinheit, bei dem ein Programm Zeit und Temperatur steuert. Der PCR-Prozess, das sog. "Amplifizieren", besteht aus mehreren (normalerweise 15-30) Wiederholungen der drei folgenden Schritte (siehe Abbildung 1):
- Melting (Schmelzen, 96°C, 30-600 Sekunden). Im Melting-Schritt wird die doppelsträngige DNA wie ein Reißverschluss in ihre beiden Einzelstränge aufgetrennt. Derzeit (April 2001) üblich ist eine thermostabile (d.h., bei hohen Temperaturen stabile) DNA-Polymerase. Diese DNA-Polymerase, die aus Bakterien aus heißen Quellen stammt, wird, im Gegensatz zu den meisten anderen Enzymen, durch den Melting-Schritt nicht deaktiviert. Diese Polymerase wird taq-Polymerase genannt, nach ihrem Ursprungsorganismus Thermophilus aquaticus.
- Annealing (Anlagerung, ca.37-65°C, 30-120 Sekunden). Im Annealing-Schritt lagern sich die Primer an die einzelnen DNA-Stränge an. Anschließend lagert sich die DNA-Polymerase an die angelagerten Primer an.
- Elongation (Verlängerung, 65-80°C, 30-120 Sekunden). Im Elongation-Schritt läuft die DNA-Polymerase an der einzelsträngigen DNA entlang, wobei sie den fehlenden zweiten Strang erzeugt.
Bei jedem Durchlauf der drei Schritte wird die DNA verdoppelt, die DNA-Menge steigt also exponentiell (mit Basis 2) an. So werden beispielsweise in 30 Durchläufen von einem DNA-Strang 230 = 1 073 741 824 exakte Kopien von dem durch die Primer bestimmten Teil angefertigt. Es ist allerdings ratsam, die Anzahl der Durchläufe zu begrenzen, weil sich mit der Zeit doch einige Fehler bei den Verdoppelungsprozessen einschleichen können.
Abbildung 1: Schematische Darstellung des PCR-Zyklus.
Die 3'- und 5'-Markierungen zeigen die Ausrichtung der DNA-Stränge. Viele Enzyme laufen nur in eine Richtung entlang eines DNA-Strangs, die DNA-Polymerase zum Beispiel nur in Richtung des 3'-Endes.
(1) Schmelzen bei 96°C. (2) Anlagerung bei 68°C. (3) Verlängerung bei 72°C (P=Polymerase). (4) Der erste Zyklus ist komplett. Die beiden erzeugten DNA-Stränge werden zum Ausgangsmaterial für den nächsten Zyklus, wodurch sich die Menge an DNA bei jedem Zyklus verdoppelt.
Durch PCR wurde es möglich, ein einzelnes Gen, oder auch nur einen Teil davon zu klonieren. Das sollte nicht mit dem Klonen ganzer Lebewesen verwechselt werden. Für die Klonierung eines Gens muss es zuerst aus einem Organismus entnommen und dann in einen anderen, z.B. ein Bakterium, eingebaut werden. Dann kann das klonierte Gen im neuen Organismus detailliert untersucht werden.
PCR wird häufig verwendet, um das Gen aus dem ersten Organismus durch Vervielfältigung zu isolieren, bevor es in den zweiten transferiert wird. PCR wird auch benutzt, um gezielt Mutationen (Veränderungen) in DNA-Stängen hervorzurufen. Ein derart verändertes Gen kann dann auf die Auswirkungen der Veränderung hin untersucht werden. Auch in der Biotechnologie wird PCR häufig verwendet, z.B. um Bakterien so zu verändern, dass sie Medikamente herstellen.
Siehe auch: Polymerase-Kettenreaktion (Langfassung)